小田 有沙 | Arisa Oda
東京大学大学院総合文化研究科 助教
研究室ホームページ
多細胞化の起源を秘める菌類の排他的コミュニケーションの解明
生物の多細胞化の起源において、単細胞生物の原始的な細胞間コミュニケーションの発達は、より高次に自己組織化された生命現象の礎となってきたであろう。だが、単細胞生物では、自身の増殖を抑制しうるような細胞間コミュニケーションはこれまでほとんど報告されていなかった。我々は、単細胞菌類である酵母が、グルコースの枯渇時に、自己と同種の細胞をも殺しうる毒を分泌しつつ、毒に耐性を獲得して、自己の子孫細胞以外の競合者となる「新参者」の増殖を抑える現象を見出してきた。このlatecomer killingの現象は、複数種の酵母で観察されるが、系統的に近いからといって必ずしも保存されるわけではない。このため、生育環境条件などにより、菌類の進化の過程で、多細胞化・単細胞化の転移が起こるとともに、latecomer killingによるコミュニケーションが獲得されたり、失われたりしたと考えられる。この環境中のリソースを独占するような生存戦略は、多細胞進化においては、自律的な増殖抑制システムとして働くと予想される。そこで、この現象を起点として、単細胞・多細胞の転移が比較的起こりやすい菌類を用い、多細胞生物の自己組織化において、競合的な細胞間コミュニケーションが果たしてきた進化的な意義の解明を目指す。このために、細胞生物学・数理生物学の両アプローチから、広範な菌類における競合的な細胞間コミュニケーションの普遍性やその作用機序を解析することで、多細胞化の進化的な意義を検証する。
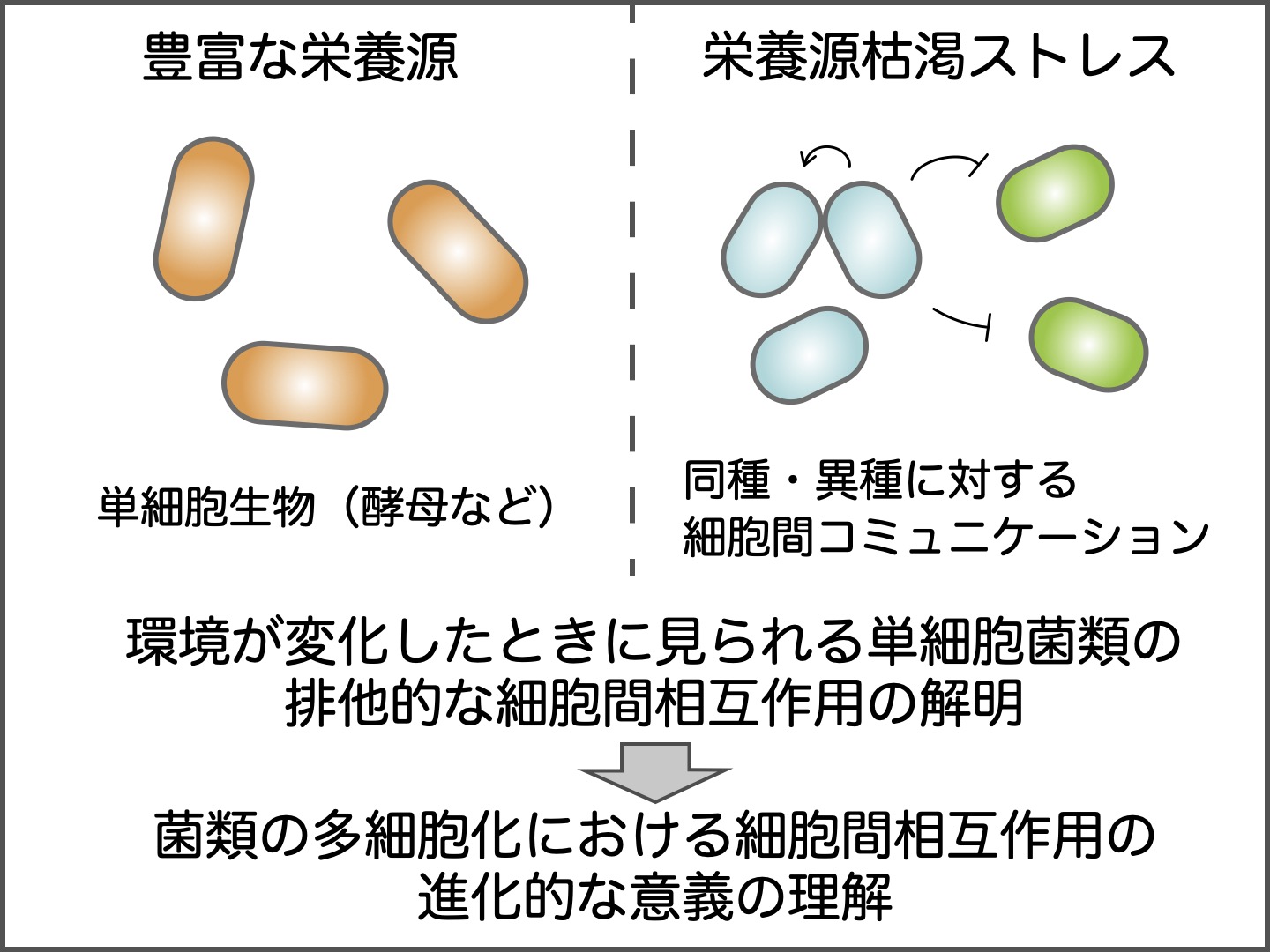
- *Oda AH, Tamura M, Kaneko K, Ohta K, *Hatakeyama TS.
Autotoxin-mediated latecomer killing in yeast communities.
PLoS Biology 20; e3001844-e3001844, 2022.
- Yasukawa T#, Oda AH#, Nakamura T, Masuo N, Tamura M, Yamasaki Y, Imura M, Yamada T, *Ohta K (# equal contribution).
TAQing2.0 for genome reorganization of asexual industrial yeasts by direct protein transfection.
Communications Biology 5; 144-144, 2022.
- *Hirata Y, Oda AH, Motono C, Shiro M, Ohta K.
Imputation-free reconstructions of three-dimensional chromosome architectures in human diploid single-cells using allele-specified contacts.
Scientific Reports 12; 2022.
- Kawashima Y, Oda AH, Hikida Y, *Ohta K.
Chromosome-dependent aneuploid formation in Spo11-less meiosis.
Genes to cells 28; 129-148, 2022.
- Muramoto N#, *Oda A#, Tanaka H#, Nakamura T#, Kugou K, Suda K, Kobayashi A, Yoneda S, Ikeuchi A, Sugimoto H, Kondo S, Ohto C, Shibata T, *Mitsukawa N, *Ohta K (# equal contribution).
Phenotypic diversification by enhanced genome restructuring after induction of multiple DNA double-strand breaks.
Nature Communications 9; 1995, 2018.
- *Hirata Y#, *Oda A#, Ohta K, Aihara K (# equal contribution).
Three-dimensional reconstruction of single-cell chromosome structure using recurrence plots.
Scientific Reports 6; 34982, 2016.
- Oda A, Takemata N, Hirata Y, Miyoshi T, Suzuki Y, Sugano S, *Ohta K.
Dynamic transition of transcription and chromatin landscape during fission yeast adaptation to glucose starvation.
Genes to Cells 20(5); 392-407, 2015.

